Biomni:通用生物医学人工智能代理
链接
https://www.biorxiv.org/content/10.1101/2025.05.30.656746v1.full
pdf:
https://biomni.stanford.edu/paper.pdf
背景
以前的方法很大程度上依赖于为缩小生物医学任务范围而定制的专业代理工作流程,这限制了它们在回答关键研究问题所需的整个生物医学领域的流畅移动和概括的能力。给人工智能代理能够处理广泛的生物医学任务带来了巨大的技术挑战。
最明显的是,需要将高级推理与执行高度专业化的生物医学动作的能力紧密结合起来。虽然基于 LLM 的推理已经取得了重大进展,但这样的 LLM 需要访问明确定义生物医学动作空间的环境,而生物医学动作空间本质上是多样化、领域特定和复杂的。此外,一个真正有能力的系统需要一个能够与这种生物医学环境进行本地交互的代理架构——自主选择和组合动作,使用其推理能力来计划和执行各种任务,而不依赖于僵化的、预定义的工作流程。
概括
Biomni,一款通用型生物医学 AI 代理,旨在自动化和推进广泛子领域的生物医学研究。Biomni 扮演着虚拟 AI 生物学家的角色,能够自主提出新颖且可验证的假设,执行复杂的生物信息学分析,并设计严谨的实验方案。
为了实现这一能力,我们首先通过系统分析从主要生物文献库中精选的涵盖 25 个不同子领域的数万篇生物医学研究论文,构建了一个统一且全面的生物医学行动空间。在此基础上,我们开发了一个由 LLM 驱动的行动发现代理,该代理能够阅读论文并提取推动生物医学发现的关键任务、工具和数据库。然后,我们筛选这些元素并将其部署到 Biomni-E1 中,Biomni-E1 是定义代理交互的生物医学行动空间的基础环境。Biomni-E1 包含 150 个专用生物医学工具、105 个软件包和 59 个数据库。
我们随后设计了Biomni-A1,这是一种通用的代理架构,能够利用Biomni E1提供的工具和数据集灵活地执行各种生物医学任务。对于用户提出的查询,该代理首先使用检索系统来识别所需的最相关工具、数据库和软件。然后,它运用基于LLM的推理和领域专业知识,生成详细的分步计划。每个步骤都通过可执行代码表达,从而实现精确灵活的生物医学操作组合——鉴于该领域对高度专业化的工具和数据资源的依赖,这一点至关重要。
与传统的函数调用方法不同,这种方法支持生物医学工作流程的动态性和复杂性。这个集成系统使Biomni不仅能够高效地解决具有挑战性的大规模生物医学问题,还能将其推广到前所未有的生物医学研究领域的新任务。
测试结果
严格的基准测试表明,Biomni 在现有的生物医学问答基准测试中表现出色,并在八个开发过程中从未遇到的具有挑战性的实际场景中表现出强大的泛化能力。此外,我们通过三个具有影响力的案例研究强调了 Biomni 的实践能力:
(1) 分析 458 个可穿戴传感器数据文件以产生新的见解;
(2) 快速对海量原始数据集(例如单细胞 RNA 测序和 ATAC 测序数据)进行全面的生物信息学分析,以产生新的见解和假设;
(3) 自主设计实验室方案以协助湿实验室研究人员。
Biomni 是我们推出的第一代可扩展通用生物医学 AI 代理,为虚拟 AI 生物学家与人类研究人员协同工作的时代奠定了基础,从而显著加速了从基础研究到转化的生物医学发现。
代理结构
Biomni 是一种通用的生物医学 AI 代理,由两个主要组件组成:
-
Biomni-E1(具有统一动作空间的
基础生物医学环境); -
Biomni-A1(旨在有效利用该环境的智能代理)。
如何整理一个统一的生物医学行动空间?
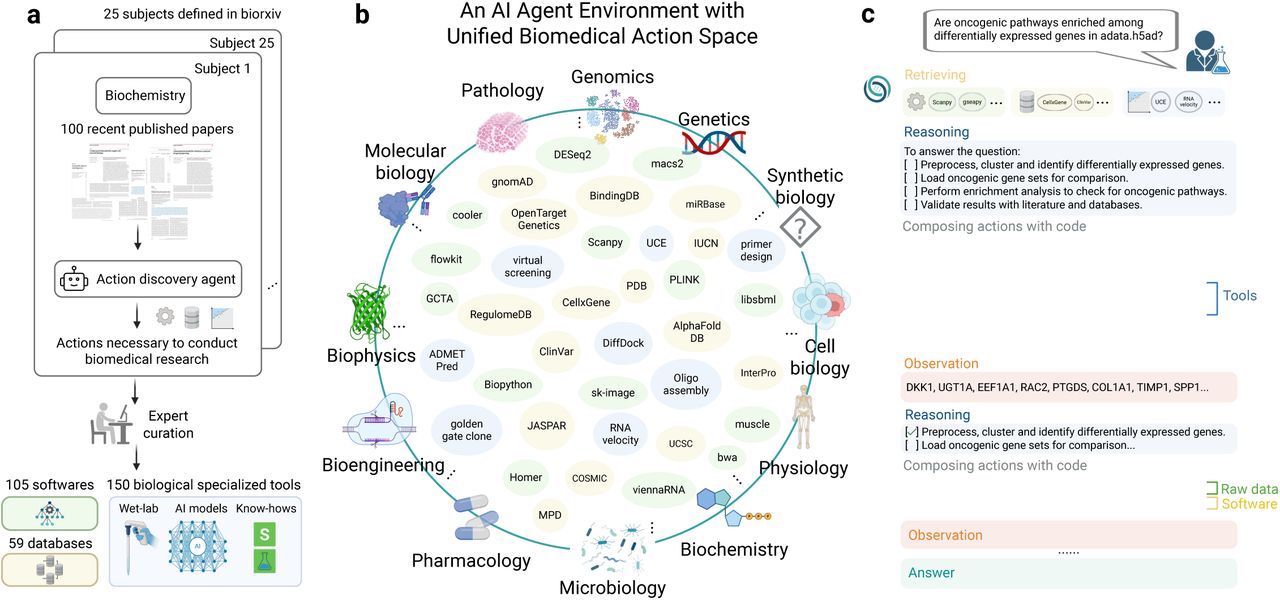
Biomni 中统一生物医学行动空间和代理环境的概览。
(a) 系统化整理统一生物医学行动空间的工作流程。使用 AI 驱动的发现代理,从 25 个生物医学子领域的 2,500 篇近期 bioRxiv 出版物中提取了开展生物医学研究所需的行动。提取的行动经过人类专家的严格验证和整理,最终整合了 105 种生物医学软件工具、150 种专用生物学工具(包括湿实验室方案、AI 驱动的预测模型和特定领域的专业知识)以及 59 个综合生物医学数据库。
(b) 涵盖遗传学、基因组学、合成生物学、细胞生物学、生理学、微生物学、药理学、生物工程、生物物理学、分子生物学和病理学等不同生物医学子领域的统一生物医学行动空间的图示。图中展示了集成到 Biomni 环境中的代表性工具和数据库,突出了其通用功能。
© 示例工作流程展示了 Biomni 的推理和动作组合过程,该过程能够自主回答一个复杂的生物学问题。Biomni 根据用户的查询检索相关工具,制定结构化的推理计划,并编写可执行代码以执行全面的生物信息学分析,并根据观察结果不断改进其推理,直至最终得到精确的答案。
如何构建一个能够处理各种生物医学任务的通用代理?
一个专门的代理架构——一种避免为每个单独任务硬编码工作流的架构。这促成了 Biomni-A1 的开发,它融合了多项对整个生物医学研究领域至关重要的核心创新。
首先,我们引入了一种基于 LLM 的工具选择机制,旨在应对生物医学工具的复杂性和专业化,并根据用户目标动态检索定制的资源子集。
其次,考虑到生物医学任务通常需要丰富的程序逻辑,Biomni-A1 使用代码作为通用操作接口——使其能够编写和执行涉及循环、并行化和条件逻辑的复杂工作流。至关重要的是,这种方法还使代理能够交错调用不符合预定义函数签名的软件、工具、数据库和原始数据操作——从而支持灵活、动态地集成异构资源。
第三,该智能体采用自适应规划策略:它基于生物医学知识制定初始计划,并在执行过程中不断迭代完善,从而实现响应迅速、情境感知的行为。
这些创新共同使 Biomni-A1 能够泛化到前所未有的任务和领域,动态地组合智能动作,并与软件、数据和工具进行交互,从而体现出通用生物医学智能(图 1c)。
效果
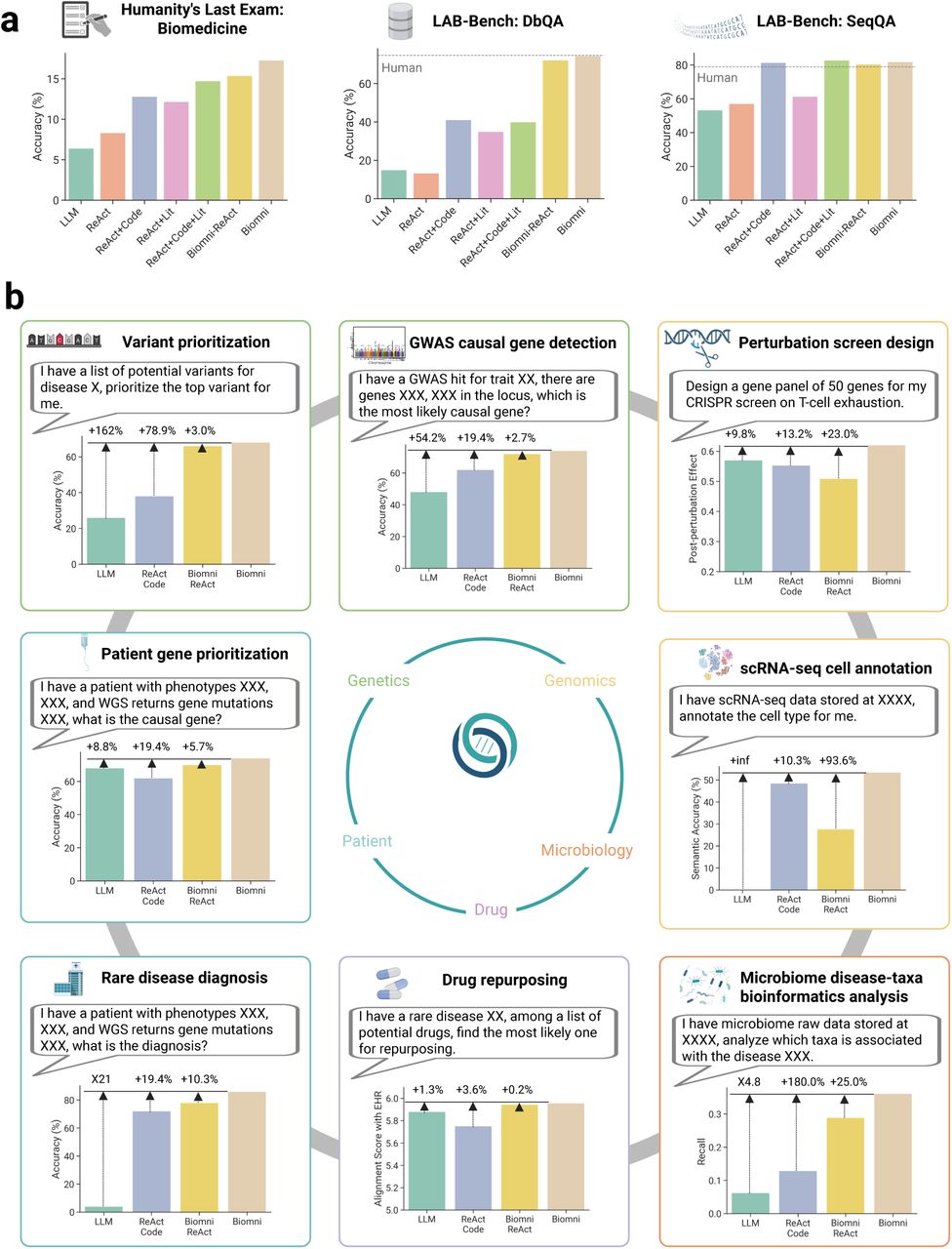
Biomni 在各种现实生物医学任务中的零样本泛化。
(a) 在问答类多项选择基准测试中,Biomni 的表现优于 6 个基准模型,这些基准测试广泛评估了该模型在生物医学领域的能力;
(b) Biomni 在八个前所未有的真实生物医学场景中展现出强大的零样本性能,这些场景涵盖多个生物医学子领域,且无需任何特定任务的微调或快速工程。评估的任务包括:
变异优先级排序(基因组学):从某个性状的潜在变异列表中找出最可能的致病变异,这需要推理非编码区域的调控功能;GWAS 致病基因检测(遗传学和基因组学):选择某个基因座内最可能的致病基因,这需要细粒度的基因座级推断;扰动筛选设计(功能基因组学和免疫学):构建基因面板,以在大型(>20,000 个基因)搜索空间中最大化扰动后效应;患者基因优先级排序:给定个体患者的基因图谱和表型描述,确定最合理的致病基因;罕见疾病诊断(临床基因组学):将患者表型和基因发现与罕见病诊断联系起来;药物再利用(药理学):给定一种罕见疾病和一系列候选药物,选择最佳治疗方案;微生物组疾病分类单元生物信息学分析(微生物学):对微生物组数据集进行统计关联检验,以发现与疾病相关的分类单元;单细胞 RNA 测序细胞注释(单细胞生物学):为跨组织、跨物种和跨平台的单个细胞谱分配准确的细胞类型标签;
在这些不同的场景中,Biomni 的表现始终优于基线模型(Base LLM、ReAct+Code)和专门环境(Biomni ReAct),突显了其通用生物医学能力以及自主适应新的复杂生物医学任务的能力。
Biomni 联合分析 458 个可穿戴传感器文件以生成生理假设
略。
Biomni 自动化复杂的多组学分析,以揭示骨骼谱系的转录调控
略。
讨论
Biomni 通过将复杂且劳动密集型的工作流程(通常需要专业知识和编程技能)自动化,使研究人员能够将精力转向创造性假设生成、实验创新和跨学科合作。这一转变意义深远。
- 在生物制药的靶点和药物发现领域,Biomni 可以自主确定靶点的优先级、设计扰动筛选或重新利用药物,从而为更快、更具成本效益的研究提供途径。
- 在临床应用领域,其在基因优先级排序和罕见病诊断方面的能力有助于提供更精准、个性化的洞察和更高效的诊断流程。
- 在消费者健康领域,Biomni 将可穿戴数据与多组学分析相结合,旨在实现实时、个性化的健康监测和干预。
局限性
- 虽然 Biomni 的统一环境涵盖了广泛的生物医学工具和数据库,但评估的任务仅代表了该领域的一个子集,关键领域仍未得到探索。
- 在行动发现代理中,我们优先考虑最新文献的决定使代理显得与时俱进,但这也存在着忽略一些基础概念和技术的风险,这些概念和技术尽管具有持久的相关性,但已经从当前的讨论中淡出。
- 尽管 Biomni 在数据库查询、序列分析和分子克隆等任务上的表现接近人类水平,但在需要细致入微的临床判断、新颖的实验推理、分析性发明或深度生物学思考与综合的领域,它仍然举步维艰。目前还没有系统能够完全涵盖人类生物医学专业知识。
方法
从文献中发现行动
收集并分析了 biorxiv 上 2024 年的 100 篇近期出版物,提取并解析其 PDF 内容。每篇论文均按块处理,并由专门的提示引导法 (LLM) 逐一识别并提取三类可操作的见解:任务、软件和数据库。
具体到任务方面,LLM 被要求重点突出那些在生物医学研究工作流程中需要专门实现的重复性任务。
实施 Biomni 环境
在环境构建的初始迭代中,我们采用了一种保守且有针对性的工具管理方法。
最初,我们根据与主要研究兴趣(药物研发和临床生物医学)的相关性筛选任务,这些研究领域涵盖生物化学、生物工程、生物物理学、癌症生物学、细胞生物学、发育生物学、遗传学、基因组学、免疫学、微生物学、分子生物学、病理学、药理学、生理学、合成生物学和系统生物学。
随后,我们将这些任务的范围缩小到大约 1,900 个常见的重复任务。我们进一步手动审查这些任务,以消除冗余,并排除那些琐碎或易于通过简单代码实现的任务。我们强调选择需要大量领域专业知识的高度专业化任务,例如湿实验室方案和高级 AI 模型。
随后,人类科学家与配备网络搜索功能的软件工程代理合作,实现了每个专用工具。每个工具都经过了严格的验证,需要一个明确定义的测试用例来确保其成功通过。这一严格的流程最终催生了150个专用工具的精选集。此外,还包含了PubMed和Google Scholar等重要的文献检索工具,并为未来的迭代扩展做好了准备。
每个工具都使用一份全面的清单进行严格定义,该清单要求:
(1)清晰且具有描述性的名称,
(2)详细的文档,
(3)格式化为针对 LLM 解释优化的详细研究日志的输出,
(4)包含并成功通过特定的测试用例,
(5)专业化标准 - 如果可以通过简短的 LLM 生成的代码(例如,简单的数据库查询)轻松实现任务,则无需创建专门的工具。
对数据库进行了分类,并使用统一的查询函数集成了可通过 Web API 访问的大量关系数据库(例如 PDB、OpenTargets、ClinVar)。该函数接受自然语言输入,并利用 LLM 动态解析数据库模式并执行相应的查询。缺少 Web API 的数据库会被下载并在本地预处理成结构化的 Pandas DataFrame,以便代理无缝访问。
在软件集成方面,考虑到经常需要同时使用多个软件工具,我们构建了一个统一的容器化环境,并预装了一套完整的相关软件。此外,该环境还支持执行 R 包和命令行界面 (CLI) 工具。
Biomni-A1
Biomni 智能体是一个基于 CodeAct 31框架构建的通用生物医学 AI 智能体,旨在通过将 LLM 与交互式编码环境相结合,系统地解决生物医学任务。
当用户提出查询时,Biomni 首先会提示 LLM 生成一个清晰的、带编号的项目符号列表计划,详细说明解决给定问题所需的步骤,并持续跟踪进度和调整。由于工具、软件和数据库空间巨大,查询任务可能仅使用其中一小部分资源。为了避免冗长的上下文,我们使用了一个基于提示的检索器,该检索器由一个独立的 LLM 驱动,智能体会从可用资源中动态选择最相关的函数、数据集和软件库。在执行过程中,LLM 会生成代码,在编码环境(Python、R 或 Bash)中执行,并返回结果观察结果以指导后续推理。这种迭代方法持续进行,直到智能体收敛到一个准确且经过验证的解决方案。

